Tổng quan
Trong thập kỷ qua, các đơn vị xử lý đồ họa hay GPU đã trở nên phổ biến trong điện toán khoa học vì khả năng khai thác mức độ song song cao của chúng. NVIDIA có một số ứng dụng khoa học đời sống được tối ưu hóa và chạy trên GPU đa năng của họ. Thật không may, những GPU này chỉ có thể được lập trình với khung CUDA, OpenACC và OpenCL. Hầu hết các thành viên của cộng đồng khoa học đời sống đều không quen thuộc với các khuôn khổ này và vì vậy rất ít nhà sinh vật học hoặc nhà tin sinh học có thể sử dụng hiệu quả các kiến trúc GPU. Tuy nhiên, GPU đã xâm nhập vào lĩnh vực mô phỏng động lực phân tử (MDS) kể từ khi MD được phát triển vào những năm 1950. MDS yêu cầu công việc tính toán nặng nề để mô phỏng các cấu trúc phân tử sinh học hoặc các tương tác của chúng.
Trong blog này, chúng tôi đã thử nghiệm hai ứng dụng MDS; NAMD và LAMMPS sử dụng máy chủ Dell EMC PowerEdge XE8545 với GPU NVIDIA A100. Vì máy chủ XE8545 không hỗ trợ GPU NVIDIA V100 nên chúng tôi có thể ước tính sơ bộ mức tăng hiệu suất với A100 từ các thử nghiệm trước đây của chúng tôi.
Hai ứng dụng này là các gói MD song song miễn phí và mã nguồn mở được thiết kế để phân tích các chuyển động vật lý của các nguyên tử và phân tử.
Cấu hình máy chủ thử nghiệm được tóm tắt trong bảng sau.
| Dell EMC PowerEdge XE8545 | |
| CPU | 2x 7713 (Milan), 64 lõi, 2,0 GHz – 3,7 GHz Base-Boost, TDP 225 W, 256 MB L3 Cache |
| ĐẬP | DDR4 1024 GB (32 x 32 GB) 3200 MT/s |
| Hệ điều hành | RHEL 8.3 (4.18.0-240.el8.x86_64) |
| Mạng hệ thống tập tin | Mellanox InfiniBand HDR100 |
| Hệ thống tập tin | Giải pháp sẵn sàng của Dell EMC cho bộ lưu trữ dung lượng cao HPC BeeGFS |
| Hồ sơ hệ thống BIOS | Tối ưu hóa hiệu suất |
| Bộ xử lý logic | Tàn tật |
| Công nghệ ảo hóa | Tàn tật |
| Máy gia tốc | 4 x A100-40GB SXM4 |
| Cuda/Bộ công cụ | 11.2 |
| OpenMPI | 4.1.1 |
| NAMD | NAMD_Git-2021-04-01_Source |
| LAMMPS | Phiên bản ổn định (29/10/2020) |
Đánh giá hiệu suất
NAMD
Động lực phân tử quy mô nano (NAMD) là phần mềm nguồn mở để mô phỏng động lực phân tử được viết bằng mô hình lập trình song song CHARMM và được thiết kế để mô phỏng hiệu suất cao của các hệ thống phân tử sinh học lớn.
NAMD được xây dựng bằng mã nguồn NAMD_Git-2021-04-01_Source trên GCC 11.1 và CUDA 11.2. Đối với các thử nghiệm của mình, chúng tôi đã sử dụng hai bộ dữ liệu; 1,06 triệu nguyên tử của hệ thống Virus khảm thuốc lá vệ tinh (STMV) và hệ thống nguyên tử HECBioSim 3000k , là một cặp tetramers 1IVO và 1NQL hEGFR.
Hình 1 cho thấy hiệu suất của GPU A100 gấp 4 lần với bộ dữ liệu STMV. NAMD sử dụng tùy chọn ++p để chỉ định số lượng luồng công việc và theo khuyến nghị, bằng tổng số lõi trừ đi tổng số GPU. Tuy nhiên, tổng số lõi trong dòng bộ xử lý Milan Eypc 7003, chẳng hạn như Eypc 7713 được sử dụng trong hệ thống thử nghiệm, không tuân theo khuyến nghị chung. Nó có vẻ là khoảng 79 đến 90 lõi. Số lượng lõi tối ưu phụ thuộc vào kích thước dữ liệu. Hiệu suất mô phỏng gần 9 nano giây (ns) mỗi ngày là mức tăng hiệu suất đáng kể từ các thử nghiệm NVIDIA V100 mà chúng tôi đã thực hiện trước đây. Khó có thể nói mức tăng hiệu suất là đóng góp duy nhất của GPU A100 mới vì việc so sánh V100 16 GB trên nền tảng Intel Skylake với 40 GB A100 trên nền tảng AMD Milan có thể không chính xác.
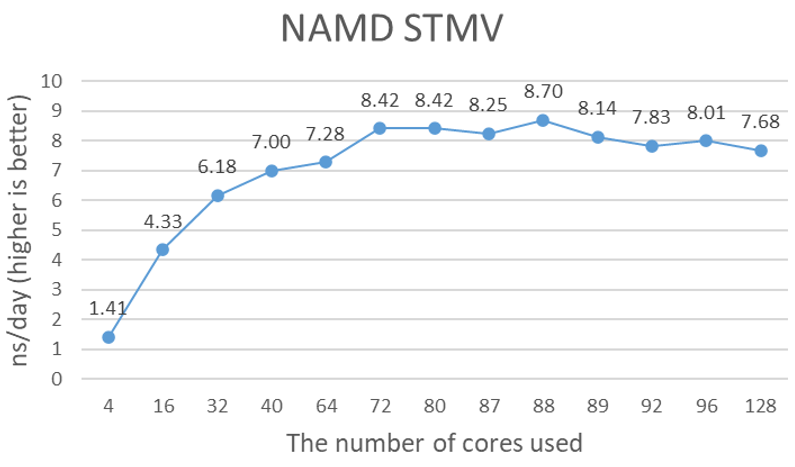
Hình 1. Thời gian mô phỏng ước tính mỗi ngày với GPU NVIDIA A100 4x
Mục đích của thử nghiệm bổ sung với 3 triệu tetramer protein nguyên tử là để xác nhận rằng kết quả thử nghiệm STMV không phải là giả tạo do cấu trúc khối 20 mặt tương đối nhỏ của SMTV và mô phỏng một phần quá trình lắp ráp và tháo rời. Hình 2 cho thấy biểu đồ mô phỏng nano giây mỗi ngày cho dữ liệu nguyên tử 3000k. 2,1 ns/ngày dường như gần đạt hiệu suất tối đa với 64 lõi.
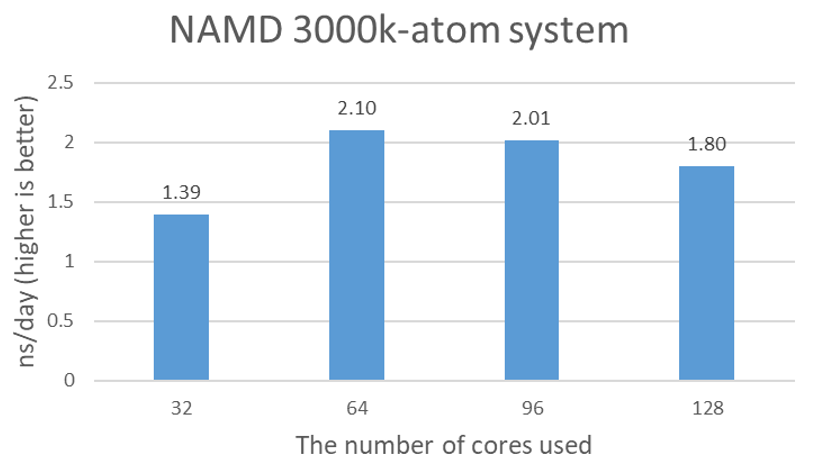
Hình 2. Thời gian mô phỏng ước tính mỗi ngày với GPU NVIDIA A100 4x
LAMMPS
Bộ mô phỏng song song nguyên tử/phân tử quy mô lớn, hay LAMMPS, là một mã động lực phân tử cổ điển và có tiềm năng cho các vật liệu trạng thái rắn (kim loại và chất bán dẫn), vật chất mềm (phân tử sinh học và polyme), và các hệ thống hạt thô hoặc siêu âm. LAMMPS có thể mô hình hóa các nguyên tử hoặc có thể được sử dụng làm trình mô phỏng hạt song song ở quy mô nguyên tử, trung bình hoặc liên tục. LAMMPS chạy trên các bộ xử lý đơn lẻ hoặc song song bằng cách sử dụng các kỹ thuật truyền thông báo và phân rã không gian của miền mô phỏng. LAMMPS được xây dựng bằng GCC 11.1, OpenMPI 4.1.1 và CUDA 11.2 từ nguồn. Hệ thống nguyên tử 465k được chọn từ HECBioSim .
Như được hiển thị trong Hình 3, LAMMPS có quy mô lớn theo số lượng A100. Với GPU A100 4x, có thể đạt được mô phỏng 8,4 ns/ngày.
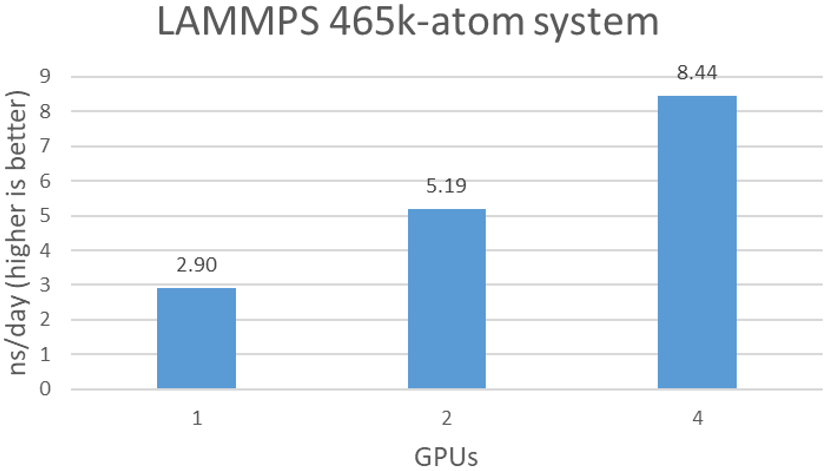
Hình 3. Thời gian mô phỏng ước tính mỗi ngày với số lượng BPU khác nhau
Phần kết luận
Mặc dù không thể so sánh hiệu suất của A100 và V100 từ nghiên cứu này, nhưng CPU Milan và A100 cho thấy sức mạnh tổng hợp mạnh mẽ giữa nhiều lõi hơn với GPU tốt hơn và nhanh hơn. Chạy NAMD và LAMMPS trên XE8545 với A100 có thể mang lại hiệu suất tốt hơn hệ thống với V100 .

Bài viết mới cập nhật
Đơn giản hóa hoạt động bảo mật cho nền tảng Dell HCI với NSX
Hầu hết các công ty công nghệ trong lĩnh vực CNTT ...
Lợi ích của cơ sở hạ tầng có thể cấu hình cho VMware Cloud Foundation trên các nút sẵn sàng vSAN
Các cuộc thảo luận so sánh kiến trúc đám mây công ...
Các nút Dell vSAN Ready với Dell ObjectScale để chạy các ứng dụng hiện đại của bạn
Tôi tập trung làm nổi bật giá trị mà các Node ...
Lợi ích của AMD dành cho các nút sẵn sàng cho Dell vSAN
Trong bài viết trước, tôi đã giải thích cách cung cấp nhiều nền ...